Oficina de análise de dados NIR em ambiente R
Partes 1—3 - Manejo de dados com dplyr, preparação de dados, e PCA
Ricardo Perdiz (Luz da Floresta) | 2021/06/30
Sobre mim

Dr. em Botânica (INPA)
Analista de dados
@ricoperdiz
@ricoperdiz
ricardoperdiz.com
ricoperdiz@gmail.com
Sumário
Uma breve introdução
Manipulação de dados com o pacote dplyr
Preparação dos dados para análise
Sumário
Uma breve introdução
Manipulação de dados com o pacote dplyr
Preparação dos dados para análise
Análise de componentes principais (PCA)
"There should be one – and preferably only one – obvious way to do it." (The Zen of Python)1
"There should be one – and preferably only one – obvious way to do it." (The Zen of Python)1
Paradigma em linguagens de programação.
"There should be one – and preferably only one – obvious way to do it." (The Zen of Python)1
Paradigma em linguagens de programação.
"There’s more than one way to do it" (Perl, R)
Há mais de uma maneira de se fazer uma determinada ação no R
plot(mtcars$hp, mtcars$mpg)plot(mpg ~ hp, data = mtcars)library(lattice)xyplot(mpg ~ hp, data = mtcars)library(ggplot2)ggplot(mtcars, aes(x = hp, y = mpg)) + geom_point()Várias maneiras de se fazer algo em R, todas com vantagens e desvantagens. O guia de desenvolvedor do Python afirma: R é muito diferente de Python nesse aspecto. Uma vantagem da diversidade de interfaces do R é que ela pode se desenvolver com o tempo e se ajustar às diferentes necessidades dos usuários.
Por que ensinar ferramentas do tidyverse, caret e tidymodels?
Por que ensinar ferramentas do tidyverse, caret e tidymodels?
Enraizar a idéia de ter UM conjunto de ferramentas para trabalhar e analisar os dados
Instruções gerais
data.frame(a = 1:10, b = 1:10)Um código por linha!
data.frame( a = 1:10, b = 1:10)Instruções gerais
Dúvidas?
Dúvidas de R básico, eu vou apontar onde descobrir a solução, mas não vou focar em resolver nesta oficina.
Instruções gerais
Dúvidas?
Dúvidas de R básico, eu vou apontar onde descobrir a solução, mas não vou focar em resolver nesta oficina.
Podem parar e perguntar!
Dados para a oficina
1. Conjunto de dados nir_data1, que acompanha o pacote NIRtools2.
Dados para a oficina
1. Conjunto de dados nir_data1, que acompanha o pacote NIRtools2.
2. Próprios dados
Perdiz (2020) Delimitação específica e filogeografia do complexo Protium aracouchini (Aubl.) Marchand (Burseraceae). Tese de doutorado. Programa de pós-graduação em Ciências Biológicas, INPA, Manaus, Amazonas, Brasil. https://repositorio.inpa.gov.br/handle/1/36948
Perdiz (2021) https://github.com/ricoperdiz/NIRtools
Vou utilizar o conjunto de dados nir_data, um subconjunto proveniente de minha tese de doutorado (Perdiz 2020), e que acompanha o pacote NIRtools (Perdiz 2021) para servir como exemplo para as análises neste tutorial.
Vale ressaltar que esse conjunto de dados é extremamente pequeno, por isso desconsidere os resultados das análises.
Ele servirá apenas de guia para abordar os procedimentos aqui abordados.
Importação dos dados nir_data1
Para ler o conjunto de dados que utilizarei nesta oficina, basta rodar o comando abaixo:
dados <- read.table("https://raw.githubusercontent.com/ricoperdiz/NIRtools/master/inst/extdata/nir_data.csv", sep = "\t", header = TRUE, as.is = TRUE)- Perdiz (2020) https://repositorio.inpa.gov.br/handle/1/36948
Importação dos dados nir_data1
Para ler o conjunto de dados que utilizarei nesta oficina, basta rodar o comando abaixo:
dados <- read.table("https://raw.githubusercontent.com/ricoperdiz/NIRtools/master/inst/extdata/nir_data.csv", sep = "\t", header = TRUE, as.is = TRUE)- Perdiz (2020) https://repositorio.inpa.gov.br/handle/1/36948
De maneira alternativa, você pode ler estes dados utilizando a função fread() do pacote data.table:
library("data.table")dados <- fread("https://raw.githubusercontent.com/ricoperdiz/NIRtools/master/inst/extdata/nir_data.csv")Importação de dados próprios
De maneira alternativa, como no exemplo anterior, você pode utilizar a função fread() do pacote data.table. Esta função não exige a especificação do separador, pois é automaticamente descoberto:
library("data.table")dados <- fread("MEUSDADOS.csv")Transformação do data.frame em um tibble1
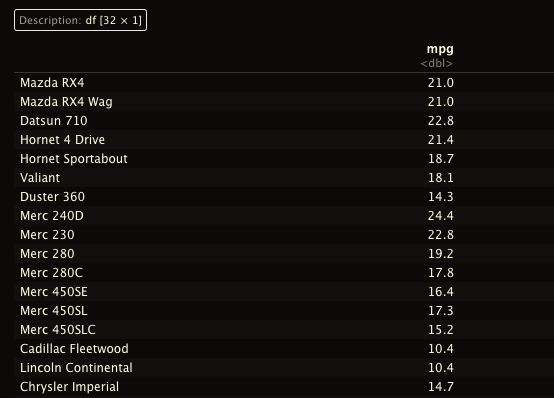
library("tibble")dados <- as_tibble(dados)head(dados)## # A tibble: 6 × 1,562## especimenid SP1 face coletor number X10001.03 X3999.64 X4003.497 X4007.354## <int> <chr> <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl>## 1 10194 P. araco… abax… Perdiz… 2856 0.278 0.596 0.596 0.596## 2 10194 P. araco… adax… Perdiz… 2856 0.254 0.567 0.567 0.567## 3 10194 P. araco… abax… Perdiz… 2856 0.273 0.581 0.582 0.582## 4 10194 P. araco… adax… Perdiz… 2856 0.258 0.576 0.577 0.577## 5 10194 P. araco… abax… Perdiz… 2856 0.278 0.596 0.596 0.596## 6 10194 P. araco… adax… Perdiz… 2856 0.254 0.567 0.567 0.567## # ℹ 1,553 more variables: X4011.211 <dbl>, X4015.068 <dbl>, X4018.925 <dbl>,## # X4022.781 <dbl>, X4026.638 <dbl>, X4030.495 <dbl>, X4034.352 <dbl>,## # X4038.209 <dbl>, X4042.066 <dbl>, X4045.923 <dbl>, X4049.78 <dbl>,## # X4053.637 <dbl>, X4057.494 <dbl>, X4061.351 <dbl>, X4065.208 <dbl>,## # X4069.065 <dbl>, X4072.922 <dbl>, X4076.779 <dbl>, X4080.635 <dbl>,## # X4084.492 <dbl>, X4088.349 <dbl>, X4092.206 <dbl>, X4096.063 <dbl>,## # X4099.92 <dbl>, X4103.777 <dbl>, X4107.634 <dbl>, X4111.491 <dbl>, …- Este passo NÃO É NECESSÁRIO. Ele facilita apenas a visualização dos dados no console, e indica o tipo de cada variável em seu conjunto de dados. Saiba mais sobre um
tibbleem https://r4ds.had.co.nz/tibbles.html.
Checagem básica
Primeiras 10 linhas e 5 colunas
dados[1:10, 1:5]## # A tibble: 10 × 5## especimenid SP1 face coletor number## <int> <chr> <chr> <chr> <int>## 1 10194 P. aracouchini abaxial Perdiz, R.O. 2856## 2 10194 P. aracouchini adaxial Perdiz, R.O. 2856## 3 10194 P. aracouchini abaxial Perdiz, R.O. 2856## 4 10194 P. aracouchini adaxial Perdiz, R.O. 2856## 5 10194 P. aracouchini abaxial Perdiz, R.O. 2856## 6 10194 P. aracouchini adaxial Perdiz, R.O. 2856## 7 10194 P. aracouchini abaxial Perdiz, R.O. 2856## 8 10194 P. aracouchini adaxial Perdiz, R.O. 2856## 9 10196 P. aracouchini abaxial Perdiz, R.O. 2858## 10 10196 P. aracouchini adaxial Perdiz, R.O. 2858Estrutura dos dados
str(dados[, 1:15])## tibble [48 × 15] (S3: tbl_df/tbl/data.frame)## $ especimenid: int [1:48] 10194 10194 10194 10194 10194 10194 10194 10194 10196 10196 ...## $ SP1 : chr [1:48] "P. aracouchini" "P. aracouchini" "P. aracouchini" "P. aracouchini" ...## $ face : chr [1:48] "abaxial" "adaxial" "abaxial" "adaxial" ...## $ coletor : chr [1:48] "Perdiz, R.O." "Perdiz, R.O." "Perdiz, R.O." "Perdiz, R.O." ...## $ number : int [1:48] 2856 2856 2856 2856 2856 2856 2856 2856 2858 2858 ...## $ X10001.03 : num [1:48] 0.278 0.254 0.273 0.258 0.278 ...## $ X3999.64 : num [1:48] 0.596 0.567 0.581 0.576 0.596 ...## $ X4003.497 : num [1:48] 0.596 0.567 0.582 0.577 0.596 ...## $ X4007.354 : num [1:48] 0.596 0.567 0.582 0.577 0.596 ...## $ X4011.211 : num [1:48] 0.596 0.567 0.582 0.577 0.596 ...## $ X4015.068 : num [1:48] 0.596 0.567 0.581 0.576 0.596 ...## $ X4018.925 : num [1:48] 0.595 0.566 0.58 0.575 0.595 ...## $ X4022.781 : num [1:48] 0.593 0.565 0.579 0.574 0.593 ...## $ X4026.638 : num [1:48] 0.592 0.563 0.578 0.573 0.592 ...## $ X4030.495 : num [1:48] 0.59 0.562 0.576 0.571 0.59 ...## - attr(*, ".internal.selfref")=<externalptr>Contagem de número de amostras por determinada categoria
SP1 é a variável que contem nome de espécie em meu conjunto de dados:
table(dados$SP1)## ## P. aracouchini P. calanense ## 24 24Plot - NIRtools1
Função nirdf()2,3 converte um objeto com dados NIR em um objeto de classe nirdf, o que permite então utilizar u método próprio para plotar em R usando a função plot().
nirdad <- NIRtools::nirdf( dados, category = "SP1", measure_columns = grep("^X", names(dados), value = TRUE), measure_columns_prefix = "X" )plot(nirdad, "SP1", cex_pt = .7, cex_leg = 2, text_font = 3)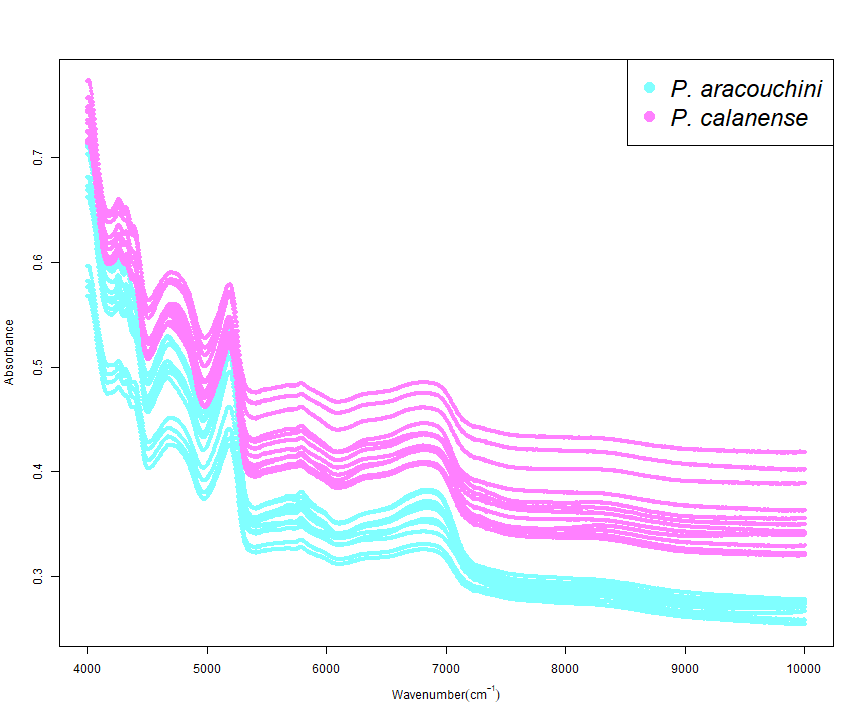
- Perdiz (2021). https://github.com/ricoperdiz/NIRtools.
Ver tutorial em https://github.com/ricoperdiz/NIRtools para aprender a usar a função
nirdad.Outra alternativa é utilizar os passos contidos em http://botanicaamazonica.wiki.br/labotam/doku.php?id=analises:nir:ver_spectra, de autoria do Dr. Alberto Vicentini (INPA).
O que é o dplyr?

Funções ou verbos para manipular e sumarizar dados tabulados.
Não é um pacote de ações rápidas.
Nomes de funções facilitam a memorização.
Nomes sem aspas dentro das funções!
Este pacote possui uma série de funções/verbos para manipular e sumarizar dados tabulados.
Não é o pacote mais rápido ou eficiente para fazer tais ações.
Porém, o uso de funções cujos nomes nos remetem às ações que efetuamos rotineiramente com dados facilita a memorização.
Somado a isso, não temos a necessidade de usar aspas nos nomes das variáveis, o que acelera a digitação.
Principais funções do pacote dplyr
- Seleção de colunas -
select()
Principais funções do pacote dplyr
Seleção de colunas -
select()Ordenar linhas -
arrange()
Principais funções do pacote dplyr
Seleção de colunas -
select()Ordenar linhas -
arrange()Filtrar linhas -
filter()
Principais funções do pacote dplyr
Seleção de colunas -
select()Ordenar linhas -
arrange()Filtrar linhas -
filter()Criar novas colunas -
mutate()
Principais funções do pacote dplyr
Seleção de colunas -
select()Ordenar linhas -
arrange()Filtrar linhas -
filter()Criar novas colunas -
mutate()Sumarizar resultados -
summarise()
Principais funções do pacote dplyr
Seleção de colunas -
select()Ordenar linhas -
arrange()Filtrar linhas -
filter()Criar novas colunas -
mutate()Sumarizar resultados -
summarise()Função especial para agrupamentos -
group_by()
Atenção na hora de carregar os pacotes
Ordem de carregar pacotes é IMPORTANTE!
library("MASS")library("dplyr")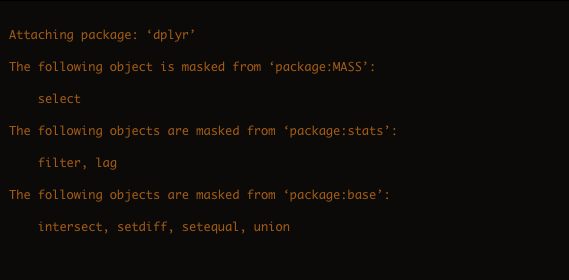
Conflito com funções de mesmo nome!
library("dplyr")library("MASS")
Selecionando colunas com select()
Podemos selecionar quantas colunas desejarmos.
Selecionando colunas com select()
Podemos selecionar quantas colunas desejarmos.
Uma coluna
select(dados, especimenid)## # A tibble: 48 × 1## especimenid## <int>## 1 10194## 2 10194## 3 10194## 4 10194## 5 10194## 6 10194## 7 10194## 8 10194## 9 10196## 10 10196## # ℹ 38 more rowsPara selecionar uma coluna, usamos a função select().
O primeiro argumento é o nome do data.frame, dados; a partir do segundo argumento, colocamos os nomes das colunas sem aspas.
Selecionando colunas com select()
Duas colunas
select(dados, especimenid, SP1)## # A tibble: 48 × 2## especimenid SP1 ## <int> <chr> ## 1 10194 P. aracouchini## 2 10194 P. aracouchini## 3 10194 P. aracouchini## 4 10194 P. aracouchini## 5 10194 P. aracouchini## 6 10194 P. aracouchini## 7 10194 P. aracouchini## 8 10194 P. aracouchini## 9 10196 P. aracouchini## 10 10196 P. aracouchini## # ℹ 38 more rowsSelecionando colunas com select()
Múltiplas colunas
select(dados, especimenid, SP1, coletor, number)## # A tibble: 48 × 4## especimenid SP1 coletor number## <int> <chr> <chr> <int>## 1 10194 P. aracouchini Perdiz, R.O. 2856## 2 10194 P. aracouchini Perdiz, R.O. 2856## 3 10194 P. aracouchini Perdiz, R.O. 2856## 4 10194 P. aracouchini Perdiz, R.O. 2856## 5 10194 P. aracouchini Perdiz, R.O. 2856## 6 10194 P. aracouchini Perdiz, R.O. 2856## 7 10194 P. aracouchini Perdiz, R.O. 2856## 8 10194 P. aracouchini Perdiz, R.O. 2856## 9 10196 P. aracouchini Perdiz, R.O. 2858## 10 10196 P. aracouchini Perdiz, R.O. 2858## # ℹ 38 more rowsRemoção de colunas
Uma coluna
dados_1colunaremovida <- select(dados, -especimenid)head(dados_1colunaremovida)## # A tibble: 6 × 1,561## SP1 face coletor number X10001.03 X3999.64 X4003.497 X4007.354 X4011.211## <chr> <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl> <dbl>## 1 P. aracouc… abax… Perdiz… 2856 0.278 0.596 0.596 0.596 0.596## 2 P. aracouc… adax… Perdiz… 2856 0.254 0.567 0.567 0.567 0.567## 3 P. aracouc… abax… Perdiz… 2856 0.273 0.581 0.582 0.582 0.582## 4 P. aracouc… adax… Perdiz… 2856 0.258 0.576 0.577 0.577 0.577## 5 P. aracouc… abax… Perdiz… 2856 0.278 0.596 0.596 0.596 0.596## 6 P. aracouc… adax… Perdiz… 2856 0.254 0.567 0.567 0.567 0.567## # ℹ 1,552 more variables: X4015.068 <dbl>, X4018.925 <dbl>, X4022.781 <dbl>,## # X4026.638 <dbl>, X4030.495 <dbl>, X4034.352 <dbl>, X4038.209 <dbl>,## # X4042.066 <dbl>, X4045.923 <dbl>, X4049.78 <dbl>, X4053.637 <dbl>,## # X4057.494 <dbl>, X4061.351 <dbl>, X4065.208 <dbl>, X4069.065 <dbl>,## # X4072.922 <dbl>, X4076.779 <dbl>, X4080.635 <dbl>, X4084.492 <dbl>,## # X4088.349 <dbl>, X4092.206 <dbl>, X4096.063 <dbl>, X4099.92 <dbl>,## # X4103.777 <dbl>, X4107.634 <dbl>, X4111.491 <dbl>, X4115.348 <dbl>, …Remoção de colunas
Duas colunas
dados_2colunasremovidas <- select(dados, -especimenid, -SP1)head(dados_2colunasremovidas)## # A tibble: 6 × 1,560## face coletor number X10001.03 X3999.64 X4003.497 X4007.354 X4011.211 X4015.068## <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>## 1 abaxial Perdiz… 2856 0.278 0.596 0.596 0.596 0.596 0.596## 2 adaxial Perdiz… 2856 0.254 0.567 0.567 0.567 0.567 0.567## 3 abaxial Perdiz… 2856 0.273 0.581 0.582 0.582 0.582 0.581## 4 adaxial Perdiz… 2856 0.258 0.576 0.577 0.577 0.577 0.576## 5 abaxial Perdiz… 2856 0.278 0.596 0.596 0.596 0.596 0.596## 6 adaxial Perdiz… 2856 0.254 0.567 0.567 0.567 0.567 0.567## # ℹ 1,551 more variables: X4018.925 <dbl>, X4022.781 <dbl>, X4026.638 <dbl>,## # X4030.495 <dbl>, X4034.352 <dbl>, X4038.209 <dbl>, X4042.066 <dbl>,## # X4045.923 <dbl>, X4049.78 <dbl>, X4053.637 <dbl>, X4057.494 <dbl>,## # X4061.351 <dbl>, X4065.208 <dbl>, X4069.065 <dbl>, X4072.922 <dbl>,## # X4076.779 <dbl>, X4080.635 <dbl>, X4084.492 <dbl>, X4088.349 <dbl>,## # X4092.206 <dbl>, X4096.063 <dbl>, X4099.92 <dbl>, X4103.777 <dbl>,## # X4107.634 <dbl>, X4111.491 <dbl>, X4115.348 <dbl>, X4119.205 <dbl>, …Remoção de colunas
Múltiplas colunas
multiplas_colunas_removidas <- select(dados, -one_of(c("especimenid", "SP1", "face")))head(multiplas_colunas_removidas)## # A tibble: 6 × 1,559## coletor number X10001.03 X3999.64 X4003.497 X4007.354 X4011.211 X4015.068## <chr> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>## 1 Perdiz, R.O. 2856 0.278 0.596 0.596 0.596 0.596 0.596## 2 Perdiz, R.O. 2856 0.254 0.567 0.567 0.567 0.567 0.567## 3 Perdiz, R.O. 2856 0.273 0.581 0.582 0.582 0.582 0.581## 4 Perdiz, R.O. 2856 0.258 0.576 0.577 0.577 0.577 0.576## 5 Perdiz, R.O. 2856 0.278 0.596 0.596 0.596 0.596 0.596## 6 Perdiz, R.O. 2856 0.254 0.567 0.567 0.567 0.567 0.567## # ℹ 1,551 more variables: X4018.925 <dbl>, X4022.781 <dbl>, X4026.638 <dbl>,## # X4030.495 <dbl>, X4034.352 <dbl>, X4038.209 <dbl>, X4042.066 <dbl>,## # X4045.923 <dbl>, X4049.78 <dbl>, X4053.637 <dbl>, X4057.494 <dbl>,## # X4061.351 <dbl>, X4065.208 <dbl>, X4069.065 <dbl>, X4072.922 <dbl>,## # X4076.779 <dbl>, X4080.635 <dbl>, X4084.492 <dbl>, X4088.349 <dbl>,## # X4092.206 <dbl>, X4096.063 <dbl>, X4099.92 <dbl>, X4103.777 <dbl>,## # X4107.634 <dbl>, X4111.491 <dbl>, X4115.348 <dbl>, X4119.205 <dbl>, …Reordenar colunas
select(dados, SP1, number, coletor)## # A tibble: 48 × 3## SP1 number coletor ## <chr> <int> <chr> ## 1 P. aracouchini 2856 Perdiz, R.O.## 2 P. aracouchini 2856 Perdiz, R.O.## 3 P. aracouchini 2856 Perdiz, R.O.## 4 P. aracouchini 2856 Perdiz, R.O.## 5 P. aracouchini 2856 Perdiz, R.O.## 6 P. aracouchini 2856 Perdiz, R.O.## 7 P. aracouchini 2856 Perdiz, R.O.## 8 P. aracouchini 2856 Perdiz, R.O.## 9 P. aracouchini 2858 Perdiz, R.O.## 10 P. aracouchini 2858 Perdiz, R.O.## # ℹ 38 more rowsReordenar colunas com everything()
select(dados, coletor, number, everything())## # A tibble: 48 × 1,562## coletor number especimenid SP1 face X10001.03 X3999.64 X4003.497 X4007.354## <chr> <int> <int> <chr> <chr> <dbl> <dbl> <dbl> <dbl>## 1 Perdiz, R… 2856 10194 P. a… abax… 0.278 0.596 0.596 0.596## 2 Perdiz, R… 2856 10194 P. a… adax… 0.254 0.567 0.567 0.567## 3 Perdiz, R… 2856 10194 P. a… abax… 0.273 0.581 0.582 0.582## 4 Perdiz, R… 2856 10194 P. a… adax… 0.258 0.576 0.577 0.577## 5 Perdiz, R… 2856 10194 P. a… abax… 0.278 0.596 0.596 0.596## 6 Perdiz, R… 2856 10194 P. a… adax… 0.254 0.567 0.567 0.567## 7 Perdiz, R… 2856 10194 P. a… abax… 0.273 0.581 0.582 0.582## 8 Perdiz, R… 2856 10194 P. a… adax… 0.258 0.576 0.577 0.577## 9 Perdiz, R… 2858 10196 P. a… abax… 0.274 0.681 0.681 0.681## 10 Perdiz, R… 2858 10196 P. a… adax… 0.278 0.673 0.672 0.672## # ℹ 38 more rows## # ℹ 1,553 more variables: X4011.211 <dbl>, X4015.068 <dbl>, X4018.925 <dbl>,## # X4022.781 <dbl>, X4026.638 <dbl>, X4030.495 <dbl>, X4034.352 <dbl>,## # X4038.209 <dbl>, X4042.066 <dbl>, X4045.923 <dbl>, X4049.78 <dbl>,## # X4053.637 <dbl>, X4057.494 <dbl>, X4061.351 <dbl>, X4065.208 <dbl>,## # X4069.065 <dbl>, X4072.922 <dbl>, X4076.779 <dbl>, X4080.635 <dbl>,## # X4084.492 <dbl>, X4088.349 <dbl>, X4092.206 <dbl>, X4096.063 <dbl>, …Funções auxiliares de select()
Há verbos que auxiliam a busca das colunas desejadas. São eles:
Funções auxiliares de select()
Há verbos que auxiliam a busca das colunas desejadas. São eles:
starts_with(): Coluna começa com um prefixo X;ends_with(): Termina com um sufixo X;contains(): Contem uma determinada palavra;matches(): Busca uma expressão regular (veja?regex);num_range(): Busca uma amplitude numérica.
starts_with()
select(dados, starts_with("col"))## # A tibble: 48 × 1## coletor ## <chr> ## 1 Perdiz, R.O.## 2 Perdiz, R.O.## 3 Perdiz, R.O.## 4 Perdiz, R.O.## 5 Perdiz, R.O.## 6 Perdiz, R.O.## 7 Perdiz, R.O.## 8 Perdiz, R.O.## 9 Perdiz, R.O.## 10 Perdiz, R.O.## # ℹ 38 more rowsends_with()
select(dados, ends_with("r"))## # A tibble: 48 × 2## coletor number## <chr> <int>## 1 Perdiz, R.O. 2856## 2 Perdiz, R.O. 2856## 3 Perdiz, R.O. 2856## 4 Perdiz, R.O. 2856## 5 Perdiz, R.O. 2856## 6 Perdiz, R.O. 2856## 7 Perdiz, R.O. 2856## 8 Perdiz, R.O. 2856## 9 Perdiz, R.O. 2858## 10 Perdiz, R.O. 2858## # ℹ 38 more rowscontains()
select(dados, contains("ol"))## # A tibble: 48 × 1## coletor ## <chr> ## 1 Perdiz, R.O.## 2 Perdiz, R.O.## 3 Perdiz, R.O.## 4 Perdiz, R.O.## 5 Perdiz, R.O.## 6 Perdiz, R.O.## 7 Perdiz, R.O.## 8 Perdiz, R.O.## 9 Perdiz, R.O.## 10 Perdiz, R.O.## # ℹ 38 more rowsmatches()
select(dados, matches("^X[0-9]+\\.97$"))## # A tibble: 48 × 1## X8438.97## <dbl>## 1 0.294## 2 0.271## 3 0.290## 4 0.275## 5 0.294## 6 0.271## 7 0.290## 8 0.275## 9 0.285## 10 0.290## # ℹ 38 more rowsnum_range()
select(dados, num_range("X", seq(from = 3999.64, to = 4007.354, by = 3.857)))## # A tibble: 48 × 3## X3999.64 X4003.497 X4007.354## <dbl> <dbl> <dbl>## 1 0.596 0.596 0.596## 2 0.567 0.567 0.567## 3 0.581 0.582 0.582## 4 0.576 0.577 0.577## 5 0.596 0.596 0.596## 6 0.567 0.567 0.567## 7 0.581 0.582 0.582## 8 0.576 0.577 0.577## 9 0.681 0.681 0.681## 10 0.673 0.672 0.672## # ℹ 38 more rowsCombinações de linhas distintas - distinct()
Ao selecionar diversas colunas, podemos querer ver combinações distintas de linhas.
Combinações de linhas distintas - distinct()
Ao selecionar diversas colunas, podemos querer ver combinações distintas de linhas.
Usamos então a função distinct() para obter essas combinações:
distinct(dados, coletor, number, SP1)## # A tibble: 6 × 3## coletor number SP1 ## <chr> <int> <chr> ## 1 Perdiz, R.O. 2856 P. aracouchini## 2 Perdiz, R.O. 2858 P. aracouchini## 3 Perdiz, R.O. 2859 P. aracouchini## 4 Perdiz, R.O. 3236 P. calanense ## 5 Perdiz, R.O. 3237 P. calanense ## 6 Perdiz, R.O. 3239 P. calanenseOrdenar linhas - arrange()
arrange(dados, number)## # A tibble: 48 × 1,562## especimenid SP1 face coletor number X10001.03 X3999.64 X4003.497 X4007.354## <int> <chr> <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl>## 1 10194 P. arac… abax… Perdiz… 2856 0.278 0.596 0.596 0.596## 2 10194 P. arac… adax… Perdiz… 2856 0.254 0.567 0.567 0.567## 3 10194 P. arac… abax… Perdiz… 2856 0.273 0.581 0.582 0.582## 4 10194 P. arac… adax… Perdiz… 2856 0.258 0.576 0.577 0.577## 5 10194 P. arac… abax… Perdiz… 2856 0.278 0.596 0.596 0.596## 6 10194 P. arac… adax… Perdiz… 2856 0.254 0.567 0.567 0.567## 7 10194 P. arac… abax… Perdiz… 2856 0.273 0.581 0.582 0.582## 8 10194 P. arac… adax… Perdiz… 2856 0.258 0.576 0.577 0.577## 9 10196 P. arac… abax… Perdiz… 2858 0.274 0.681 0.681 0.681## 10 10196 P. arac… adax… Perdiz… 2858 0.278 0.673 0.672 0.672## # ℹ 38 more rows## # ℹ 1,553 more variables: X4011.211 <dbl>, X4015.068 <dbl>, X4018.925 <dbl>,## # X4022.781 <dbl>, X4026.638 <dbl>, X4030.495 <dbl>, X4034.352 <dbl>,## # X4038.209 <dbl>, X4042.066 <dbl>, X4045.923 <dbl>, X4049.78 <dbl>,## # X4053.637 <dbl>, X4057.494 <dbl>, X4061.351 <dbl>, X4065.208 <dbl>,## # X4069.065 <dbl>, X4072.922 <dbl>, X4076.779 <dbl>, X4080.635 <dbl>,## # X4084.492 <dbl>, X4088.349 <dbl>, X4092.206 <dbl>, X4096.063 <dbl>, …Ordenar linhas - arrange()
arrange(dados, desc(number))## # A tibble: 48 × 1,562## especimenid SP1 face coletor number X10001.03 X3999.64 X4003.497 X4007.354## <int> <chr> <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl>## 1 12650 P. cala… abax… Perdiz… 3239 0.349 0.733 0.733 0.733## 2 12650 P. cala… adax… Perdiz… 3239 0.342 0.744 0.744 0.744## 3 12650 P. cala… abax… Perdiz… 3239 0.342 0.713 0.714 0.714## 4 12650 P. cala… adax… Perdiz… 3239 0.355 0.773 0.773 0.773## 5 12650 P. cala… abax… Perdiz… 3239 0.349 0.733 0.733 0.733## 6 12650 P. cala… adax… Perdiz… 3239 0.342 0.744 0.744 0.744## 7 12650 P. cala… abax… Perdiz… 3239 0.342 0.713 0.714 0.714## 8 12650 P. cala… adax… Perdiz… 3239 0.355 0.773 0.773 0.773## 9 12648 P. cala… abax… Perdiz… 3237 0.402 0.745 0.746 0.745## 10 12648 P. cala… adax… Perdiz… 3237 0.389 0.757 0.757 0.757## # ℹ 38 more rows## # ℹ 1,553 more variables: X4011.211 <dbl>, X4015.068 <dbl>, X4018.925 <dbl>,## # X4022.781 <dbl>, X4026.638 <dbl>, X4030.495 <dbl>, X4034.352 <dbl>,## # X4038.209 <dbl>, X4042.066 <dbl>, X4045.923 <dbl>, X4049.78 <dbl>,## # X4053.637 <dbl>, X4057.494 <dbl>, X4061.351 <dbl>, X4065.208 <dbl>,## # X4069.065 <dbl>, X4072.922 <dbl>, X4076.779 <dbl>, X4080.635 <dbl>,## # X4084.492 <dbl>, X4088.349 <dbl>, X4092.206 <dbl>, X4096.063 <dbl>, …Filtragem de linhas - filter()
filter(dados, SP1 == "P. calanense")## # A tibble: 24 × 1,562## especimenid SP1 face coletor number X10001.03 X3999.64 X4003.497 X4007.354## <int> <chr> <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl>## 1 12647 P. cala… abax… Perdiz… 3236 0.322 0.717 0.717 0.717## 2 12647 P. cala… adax… Perdiz… 3236 0.329 0.714 0.715 0.715## 3 12647 P. cala… abax… Perdiz… 3236 0.340 0.724 0.724 0.724## 4 12647 P. cala… adax… Perdiz… 3236 0.320 0.735 0.736 0.736## 5 12647 P. cala… abax… Perdiz… 3236 0.322 0.717 0.717 0.717## 6 12647 P. cala… adax… Perdiz… 3236 0.329 0.714 0.715 0.715## 7 12647 P. cala… abax… Perdiz… 3236 0.340 0.724 0.724 0.724## 8 12647 P. cala… adax… Perdiz… 3236 0.320 0.735 0.736 0.736## 9 12648 P. cala… abax… Perdiz… 3237 0.402 0.745 0.746 0.745## 10 12648 P. cala… adax… Perdiz… 3237 0.389 0.757 0.757 0.757## # ℹ 14 more rows## # ℹ 1,553 more variables: X4011.211 <dbl>, X4015.068 <dbl>, X4018.925 <dbl>,## # X4022.781 <dbl>, X4026.638 <dbl>, X4030.495 <dbl>, X4034.352 <dbl>,## # X4038.209 <dbl>, X4042.066 <dbl>, X4045.923 <dbl>, X4049.78 <dbl>,## # X4053.637 <dbl>, X4057.494 <dbl>, X4061.351 <dbl>, X4065.208 <dbl>,## # X4069.065 <dbl>, X4072.922 <dbl>, X4076.779 <dbl>, X4080.635 <dbl>,## # X4084.492 <dbl>, X4088.349 <dbl>, X4092.206 <dbl>, X4096.063 <dbl>, …Filtragem composta
filter(dados, SP1 == "P. calanense", number == 3237)## # A tibble: 8 × 1,562## especimenid SP1 face coletor number X10001.03 X3999.64 X4003.497 X4007.354## <int> <chr> <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl>## 1 12648 P. calan… abax… Perdiz… 3237 0.402 0.745 0.746 0.745## 2 12648 P. calan… adax… Perdiz… 3237 0.389 0.757 0.757 0.757## 3 12648 P. calan… abax… Perdiz… 3237 0.418 0.748 0.749 0.749## 4 12648 P. calan… adax… Perdiz… 3237 0.363 0.725 0.725 0.725## 5 12648 P. calan… abax… Perdiz… 3237 0.402 0.745 0.746 0.745## 6 12648 P. calan… adax… Perdiz… 3237 0.389 0.757 0.757 0.757## 7 12648 P. calan… abax… Perdiz… 3237 0.418 0.748 0.749 0.749## 8 12648 P. calan… adax… Perdiz… 3237 0.363 0.725 0.725 0.725## # ℹ 1,553 more variables: X4011.211 <dbl>, X4015.068 <dbl>, X4018.925 <dbl>,## # X4022.781 <dbl>, X4026.638 <dbl>, X4030.495 <dbl>, X4034.352 <dbl>,## # X4038.209 <dbl>, X4042.066 <dbl>, X4045.923 <dbl>, X4049.78 <dbl>,## # X4053.637 <dbl>, X4057.494 <dbl>, X4061.351 <dbl>, X4065.208 <dbl>,## # X4069.065 <dbl>, X4072.922 <dbl>, X4076.779 <dbl>, X4080.635 <dbl>,## # X4084.492 <dbl>, X4088.349 <dbl>, X4092.206 <dbl>, X4096.063 <dbl>,## # X4099.92 <dbl>, X4103.777 <dbl>, X4107.634 <dbl>, X4111.491 <dbl>, …Filtragem composta
> filter(dados, SP1 == "P. calanense", number == 3237)
Filtragem composta
> filter(dados, SP1 == "P. calanense", number == 3237)
É o mesmo que
Filtragem composta
> filter(dados, SP1 == "P. calanense", number == 3237)
É o mesmo que
> filter(dados, SP1 == "P. calanense" & number == 3237)
Filtragem composta
> filter(dados, SP1 == "P. calanense", number == 3237)
É o mesmo que
> filter(dados, SP1 == "P. calanense" & number == 3237)
Vírgula , em filter() equivale a &
Criar novas colunas - mutate()
nova_coluna <- mutate(dados, calanense = ifelse(SP1 == "P. calanense", 1, 0))select(nova_coluna, SP1, calanense)## # A tibble: 48 × 2## SP1 calanense## <chr> <dbl>## 1 P. aracouchini 0## 2 P. aracouchini 0## 3 P. aracouchini 0## 4 P. aracouchini 0## 5 P. aracouchini 0## 6 P. aracouchini 0## 7 P. aracouchini 0## 8 P. aracouchini 0## 9 P. aracouchini 0## 10 P. aracouchini 0## # ℹ 38 more rowsSumarizar resultados - summarise()
summarise(dados, media_X8438.97 = mean(X8438.97))## # A tibble: 1 × 1## media_X8438.97## <dbl>## 1 0.327A função summarise() colapsa um data.frame em uma única linha.
No entanto, a verdadeira utilidade dessa função se manifesta ao utilizarmos a função group_by para destacar os agrupamentos que desejamos fazer dentro dos dados.
Agrupamentos - group_by
dados_agrupados <- group_by(dados, SP1)dados_agrupados[1:5, 1:10]## # A tibble: 5 × 10## # Groups: SP1 [1]## especimenid SP1 face coletor number X10001.03 X3999.64 X4003.497 X4007.354## <int> <chr> <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl>## 1 10194 P. araco… abax… Perdiz… 2856 0.278 0.596 0.596 0.596## 2 10194 P. araco… adax… Perdiz… 2856 0.254 0.567 0.567 0.567## 3 10194 P. araco… abax… Perdiz… 2856 0.273 0.581 0.582 0.582## 4 10194 P. araco… adax… Perdiz… 2856 0.258 0.576 0.577 0.577## 5 10194 P. araco… abax… Perdiz… 2856 0.278 0.596 0.596 0.596## # ℹ 1 more variable: X4011.211 <dbl>Agrupamentos - group_by
dados_agrupados <- group_by(dados, SP1)dados_agrupados[1:5, 1:10]## # A tibble: 5 × 10## # Groups: SP1 [1]## especimenid SP1 face coletor number X10001.03 X3999.64 X4003.497 X4007.354## <int> <chr> <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl>## 1 10194 P. araco… abax… Perdiz… 2856 0.278 0.596 0.596 0.596## 2 10194 P. araco… adax… Perdiz… 2856 0.254 0.567 0.567 0.567## 3 10194 P. araco… abax… Perdiz… 2856 0.273 0.581 0.582 0.582## 4 10194 P. araco… adax… Perdiz… 2856 0.258 0.576 0.577 0.577## 5 10194 P. araco… abax… Perdiz… 2856 0.278 0.596 0.596 0.596## # ℹ 1 more variable: X4011.211 <dbl>Os dados estão agrupados?
is_grouped_df(dados_agrupados)## [1] TRUESuponhemos que eu queira ver médias de determinada variável para as espécies de meu conjunto de dados.
Usamos a função group_by para agrupar os dados em torno da variável SP1.
À primeira vista, não há resultado aparente.
Porém, ao usarmos essa função is_grouped_df(), temos a certeza de que os dados estão agrupados.
Não se preocupem em aprender essa função.
Foquem na lição a seguir.
Agrupa e sumariza!
summarise(dados_agrupados, media_X8438.97 = mean(X8438.97))## # A tibble: 2 × 2## SP1 media_X8438.97## <chr> <dbl>## 1 P. aracouchini 0.285## 2 P. calanense 0.369Vamos sumarizar os dados para a variável X8438.97.
Sumarizar várias colunas
dados_agrupados2 <- group_by( select(dados, SP1, ends_with("7")), SP1 )summarise_all(dados_agrupados2, .funs = mean)## # A tibble: 2 × 172## SP1 X4003.497 X4053.637 X4103.777 X4153.917 X4204.057 X4238.77 X4254.197## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>## 1 P. aracouch… 0.651 0.626 0.576 0.547 0.544 0.546 0.551## 2 P. calanense 0.736 0.709 0.653 0.622 0.620 0.625 0.629## # ℹ 164 more variables: X4292.767 <dbl>, X4304.337 <dbl>, X4342.907 <dbl>,## # X4393.047 <dbl>, X4443.187 <dbl>, X4489.47 <dbl>, X4493.327 <dbl>,## # X4543.467 <dbl>, X4582.037 <dbl>, X4593.607 <dbl>, X4632.177 <dbl>,## # X4682.317 <dbl>, X4732.457 <dbl>, X4782.597 <dbl>, X4821.167 <dbl>,## # X4832.737 <dbl>, X4871.307 <dbl>, X4882.877 <dbl>, X4921.447 <dbl>,## # X4971.587 <dbl>, X5017.87 <dbl>, X5021.727 <dbl>, X5071.867 <dbl>,## # X5110.437 <dbl>, X5122.007 <dbl>, X5160.577 <dbl>, X5172.147 <dbl>, …Combinando operações com o pipe (cano!)
Ferramenta existente em outras linguagens (bash, F#).
Combinando operações com o pipe (cano!)
Ferramenta existente em outras linguagens (bash, F#).
Simplifica códigos.
Combinando operações com o pipe (cano!)
Ferramenta existente em outras linguagens (bash, F#).
Simplifica códigos.
Facilita a leitura.
Combinando operações com o pipe (cano!)
Ferramenta existente em outras linguagens (bash, F#).
Simplifica códigos.
Facilita a leitura.
Reduz a quantidade de objetos na área de trabalho ls().
Combinando operações com o pipe (cano!)
Ferramenta existente em outras linguagens (bash, F#).
Simplifica códigos.
Facilita a leitura.
Reduz a quantidade de objetos na área de trabalho ls().
Duas opções:
%>%(pacote magrittr)|>(a partir do R 4.1).
Código sem encadeamento
summarise_all(group_by(filter(select(dados, SP1, ends_with("7")), SP1 == "P. calanense"), SP1), .funs = mean)## # A tibble: 1 × 172## SP1 X4003.497 X4053.637 X4103.777 X4153.917 X4204.057 X4238.77 X4254.197## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>## 1 P. calanense 0.736 0.709 0.653 0.622 0.620 0.625 0.629## # ℹ 164 more variables: X4292.767 <dbl>, X4304.337 <dbl>, X4342.907 <dbl>,## # X4393.047 <dbl>, X4443.187 <dbl>, X4489.47 <dbl>, X4493.327 <dbl>,## # X4543.467 <dbl>, X4582.037 <dbl>, X4593.607 <dbl>, X4632.177 <dbl>,## # X4682.317 <dbl>, X4732.457 <dbl>, X4782.597 <dbl>, X4821.167 <dbl>,## # X4832.737 <dbl>, X4871.307 <dbl>, X4882.877 <dbl>, X4921.447 <dbl>,## # X4971.587 <dbl>, X5017.87 <dbl>, X5021.727 <dbl>, X5071.867 <dbl>,## # X5110.437 <dbl>, X5122.007 <dbl>, X5160.577 <dbl>, X5172.147 <dbl>, …Possível solução - criação de objetos intermediários
dados_selecionados <- select(dados, SP1, ends_with("7"))dados_filtrados <- filter(dados_selecionados, SP1 == "P. calanense")dados_agrupados3 <- group_by(dados_filtrados, SP1)summarise_all(dados_agrupados3, .funs = mean)## # A tibble: 1 × 172## SP1 X4003.497 X4053.637 X4103.777 X4153.917 X4204.057 X4238.77 X4254.197## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>## 1 P. calanense 0.736 0.709 0.653 0.622 0.620 0.625 0.629## # ℹ 164 more variables: X4292.767 <dbl>, X4304.337 <dbl>, X4342.907 <dbl>,## # X4393.047 <dbl>, X4443.187 <dbl>, X4489.47 <dbl>, X4493.327 <dbl>,## # X4543.467 <dbl>, X4582.037 <dbl>, X4593.607 <dbl>, X4632.177 <dbl>,## # X4682.317 <dbl>, X4732.457 <dbl>, X4782.597 <dbl>, X4821.167 <dbl>,## # X4832.737 <dbl>, X4871.307 <dbl>, X4882.877 <dbl>, X4921.447 <dbl>,## # X4971.587 <dbl>, X5017.87 <dbl>, X5021.727 <dbl>, X5071.867 <dbl>,## # X5110.437 <dbl>, X5122.007 <dbl>, X5160.577 <dbl>, X5172.147 <dbl>, …Porém, a criação de objetos intermediários "suja" a área de trabalho com objetos que, muitas vezes, não chegarão a serem utilizados novamente
Vamos encadear com %>%
select(dados, SP1, ends_with("7")) %>% filter(., SP1 == "P. calanense") %>% group_by(., SP1) %>% summarise_all(., .funs = mean)## # A tibble: 1 × 172## SP1 X4003.497 X4053.637 X4103.777 X4153.917 X4204.057 X4238.77 X4254.197## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>## 1 P. calanense 0.736 0.709 0.653 0.622 0.620 0.625 0.629## # ℹ 164 more variables: X4292.767 <dbl>, X4304.337 <dbl>, X4342.907 <dbl>,## # X4393.047 <dbl>, X4443.187 <dbl>, X4489.47 <dbl>, X4493.327 <dbl>,## # X4543.467 <dbl>, X4582.037 <dbl>, X4593.607 <dbl>, X4632.177 <dbl>,## # X4682.317 <dbl>, X4732.457 <dbl>, X4782.597 <dbl>, X4821.167 <dbl>,## # X4832.737 <dbl>, X4871.307 <dbl>, X4882.877 <dbl>, X4921.447 <dbl>,## # X4971.587 <dbl>, X5017.87 <dbl>, X5021.727 <dbl>, X5071.867 <dbl>,## # X5110.437 <dbl>, X5122.007 <dbl>, X5160.577 <dbl>, X5172.147 <dbl>, …Aplicações de agrupamentos em dados NIR
Suponha que você deseja obter valores de média de cada variável NIR por indivíduo.
Aplicações de agrupamentos em dados NIR
Suponha que você deseja obter valores de média de cada variável NIR por indivíduo.
No conjunto de dados exemplo desta oficina, dados, indivíduos são reconhecidos pela variável especimenid.
Aplicações de agrupamentos em dados NIR
Suponha que você deseja obter valores de média de cada variável NIR por indivíduo.
No conjunto de dados exemplo desta oficina, dados, indivíduos são reconhecidos pela variável especimenid.
Logo, esta variável, especimenid, será nosso agrupamento.
Média por indivíduo
Sem encadeamento (sem uso do operador %>%)
summarise_all( group_by(select(dados, especimenid, SP1, starts_with("X")), especimenid, SP1), .funs = mean)## # A tibble: 6 × 1,559## # Groups: especimenid [6]## especimenid SP1 X10001.03 X3999.64 X4003.497 X4007.354 X4011.211 X4015.068## <int> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>## 1 10194 P. aracou… 0.266 0.580 0.581 0.580 0.580 0.580## 2 10196 P. aracou… 0.274 0.692 0.692 0.692 0.691 0.690## 3 10197 P. aracou… 0.274 0.680 0.680 0.680 0.680 0.679## 4 12647 P. calane… 0.328 0.723 0.723 0.723 0.723 0.722## 5 12648 P. calane… 0.393 0.744 0.744 0.744 0.744 0.744## 6 12650 P. calane… 0.347 0.741 0.741 0.741 0.741 0.741## # ℹ 1,551 more variables: X4018.925 <dbl>, X4022.781 <dbl>, X4026.638 <dbl>,## # X4030.495 <dbl>, X4034.352 <dbl>, X4038.209 <dbl>, X4042.066 <dbl>,## # X4045.923 <dbl>, X4049.78 <dbl>, X4053.637 <dbl>, X4057.494 <dbl>,## # X4061.351 <dbl>, X4065.208 <dbl>, X4069.065 <dbl>, X4072.922 <dbl>,## # X4076.779 <dbl>, X4080.635 <dbl>, X4084.492 <dbl>, X4088.349 <dbl>,## # X4092.206 <dbl>, X4096.063 <dbl>, X4099.92 <dbl>, X4103.777 <dbl>,## # X4107.634 <dbl>, X4111.491 <dbl>, X4115.348 <dbl>, X4119.205 <dbl>, …Média por indivíduo
Com encadeamento, utilizando o operador %>%
Mesmo cálculo do slide anterior
dados %>% select(., especimenid, SP1, starts_with("X")) %>% group_by(., especimenid, SP1) %>% summarise_all(., .funs = mean)## # A tibble: 6 × 1,559## # Groups: especimenid [6]## especimenid SP1 X10001.03 X3999.64 X4003.497 X4007.354 X4011.211 X4015.068## <int> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>## 1 10194 P. aracou… 0.266 0.580 0.581 0.580 0.580 0.580## 2 10196 P. aracou… 0.274 0.692 0.692 0.692 0.691 0.690## 3 10197 P. aracou… 0.274 0.680 0.680 0.680 0.680 0.679## 4 12647 P. calane… 0.328 0.723 0.723 0.723 0.723 0.722## 5 12648 P. calane… 0.393 0.744 0.744 0.744 0.744 0.744## 6 12650 P. calane… 0.347 0.741 0.741 0.741 0.741 0.741## # ℹ 1,551 more variables: X4018.925 <dbl>, X4022.781 <dbl>, X4026.638 <dbl>,## # X4030.495 <dbl>, X4034.352 <dbl>, X4038.209 <dbl>, X4042.066 <dbl>,## # X4045.923 <dbl>, X4049.78 <dbl>, X4053.637 <dbl>, X4057.494 <dbl>,## # X4061.351 <dbl>, X4065.208 <dbl>, X4069.065 <dbl>, X4072.922 <dbl>,## # X4076.779 <dbl>, X4080.635 <dbl>, X4084.492 <dbl>, X4088.349 <dbl>,## # X4092.206 <dbl>, X4096.063 <dbl>, X4099.92 <dbl>, X4103.777 <dbl>,## # X4107.634 <dbl>, X4111.491 <dbl>, X4115.348 <dbl>, X4119.205 <dbl>, …Média por indíviduo + face do folíolo
Sem encadeamento (sem uso do operador %>%)
summarise_all( group_by(select(dados, especimenid, SP1, face, starts_with("X")), especimenid, SP1, face), .funs = mean)## # A tibble: 12 × 1,560## # Groups: especimenid, SP1 [6]## especimenid SP1 face X10001.03 X3999.64 X4003.497 X4007.354 X4011.211## <int> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>## 1 10194 P. aracouchi… abax… 0.275 0.589 0.589 0.589 0.589## 2 10194 P. aracouchi… adax… 0.256 0.572 0.572 0.572 0.572## 3 10196 P. aracouchi… abax… 0.275 0.692 0.692 0.692 0.691## 4 10196 P. aracouchi… adax… 0.272 0.692 0.692 0.692 0.691## 5 10197 P. aracouchi… abax… 0.273 0.675 0.675 0.675 0.674## 6 10197 P. aracouchi… adax… 0.274 0.686 0.686 0.685 0.685## 7 12647 P. calanense abax… 0.331 0.720 0.721 0.720 0.720## 8 12647 P. calanense adax… 0.324 0.725 0.725 0.725 0.725## 9 12648 P. calanense abax… 0.410 0.747 0.747 0.747 0.747## 10 12648 P. calanense adax… 0.376 0.741 0.741 0.741 0.741## 11 12650 P. calanense abax… 0.346 0.723 0.723 0.724 0.723## 12 12650 P. calanense adax… 0.349 0.758 0.759 0.759 0.759## # ℹ 1,552 more variables: X4015.068 <dbl>, X4018.925 <dbl>, X4022.781 <dbl>,## # X4026.638 <dbl>, X4030.495 <dbl>, X4034.352 <dbl>, X4038.209 <dbl>,## # X4042.066 <dbl>, X4045.923 <dbl>, X4049.78 <dbl>, X4053.637 <dbl>,## # X4057.494 <dbl>, X4061.351 <dbl>, X4065.208 <dbl>, X4069.065 <dbl>,## # X4072.922 <dbl>, X4076.779 <dbl>, X4080.635 <dbl>, X4084.492 <dbl>,## # X4088.349 <dbl>, X4092.206 <dbl>, X4096.063 <dbl>, X4099.92 <dbl>,## # X4103.777 <dbl>, X4107.634 <dbl>, X4111.491 <dbl>, X4115.348 <dbl>, …Média por indíviduo + face do folíolo
Com encadeamento, utilizando o operador %>%
Mesmo cálculo do slide anterior
dados %>% select(., especimenid, SP1, face, starts_with("X")) %>% group_by(., especimenid, SP1, face) %>% summarise_all(., .funs = mean)## # A tibble: 12 × 1,560## # Groups: especimenid, SP1 [6]## especimenid SP1 face X10001.03 X3999.64 X4003.497 X4007.354 X4011.211## <int> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>## 1 10194 P. aracouchi… abax… 0.275 0.589 0.589 0.589 0.589## 2 10194 P. aracouchi… adax… 0.256 0.572 0.572 0.572 0.572## 3 10196 P. aracouchi… abax… 0.275 0.692 0.692 0.692 0.691## 4 10196 P. aracouchi… adax… 0.272 0.692 0.692 0.692 0.691## 5 10197 P. aracouchi… abax… 0.273 0.675 0.675 0.675 0.674## 6 10197 P. aracouchi… adax… 0.274 0.686 0.686 0.685 0.685## 7 12647 P. calanense abax… 0.331 0.720 0.721 0.720 0.720## 8 12647 P. calanense adax… 0.324 0.725 0.725 0.725 0.725## 9 12648 P. calanense abax… 0.410 0.747 0.747 0.747 0.747## 10 12648 P. calanense adax… 0.376 0.741 0.741 0.741 0.741## 11 12650 P. calanense abax… 0.346 0.723 0.723 0.724 0.723## 12 12650 P. calanense adax… 0.349 0.758 0.759 0.759 0.759## # ℹ 1,552 more variables: X4015.068 <dbl>, X4018.925 <dbl>, X4022.781 <dbl>,## # X4026.638 <dbl>, X4030.495 <dbl>, X4034.352 <dbl>, X4038.209 <dbl>,## # X4042.066 <dbl>, X4045.923 <dbl>, X4049.78 <dbl>, X4053.637 <dbl>,## # X4057.494 <dbl>, X4061.351 <dbl>, X4065.208 <dbl>, X4069.065 <dbl>,## # X4072.922 <dbl>, X4076.779 <dbl>, X4080.635 <dbl>, X4084.492 <dbl>,## # X4088.349 <dbl>, X4092.206 <dbl>, X4096.063 <dbl>, X4099.92 <dbl>,## # X4103.777 <dbl>, X4107.634 <dbl>, X4111.491 <dbl>, X4115.348 <dbl>, …Pausa para o café e PERGUNTAS

Pré-análise
Divisão de dados em conjuntos treino e teste
Criação de receitas de dados para pré-processamento dos dados
Vamos utilizar aqui as funcionalidades dos pacotes dentro da interface tidymodels (Kuhn et al. 2020).
rsample::initial_split() (Silge et al. 2021)
library("rsample") # initial_splitdados_split <- initial_split(dados, prop = 3/4)dados_split## <Training/Testing/Total>## <36/12/48>rsample::initial_split(), que é carregado automaticamente ao se chamar o pacote tidymodels - esta função cria índices para separar o conjunto de dados em treino e teste. Por padrão, a função divide o conjunto de dados em 75% para treino e 25% para teste. Caso você queira outra proporção, altere o argumento prop. Em caso de dúvida, veja o ? desta função executando o comando ?initial_split.
Divisão de dados proporcionalmente em relação a uma variável
Divisão de dados proporcionalmente em relação a uma variável
dados_split <- initial_split(dados, prop = 3/4, strata = "SP1")Conjunto treino rsample::training() vs. teste rsample::testing()
Conjunto treino rsample::training() vs. teste rsample::testing()
Conjunto treino
treino <- training(dados_split)table(treino$SP1)## ## P. aracouchini P. calanense ## 18 18ambas do mesmo pacote rsample - ambas recebem o objeto criado com a função initial_split(), dados_split, e dividem o treino em conjuntos treino e teste, respectivamente:
Conjunto treino rsample::training() vs. teste rsample::testing()
Conjunto teste
teste <- testing(dados_split)table(teste$SP1)## ## P. aracouchini P. calanense ## 6 6Reprodutibilidade na aleatorização
set.seed()
set.seed(221015)iris_split <- initial_split(iris, prop = 3/4, strata = "Species")iris_split## <Training/Testing/Total>## <111/39/150>Vamos estabelecer uma seed para que vocês cheguem ao mesmo resultado, caso utilizem estas "receitas" durante o aprendizado do tutorial.
Recomendo que vocês executem esses comandos juntamente comigo para que vocês tenham ciência de que a quebra dos dados será igual para todos.
Reprodutibilidade na aleatorização
head(training(iris_split), 20)## Sepal.Length Sepal.Width Petal.Length Petal.Width Species## 1 5.1 3.5 1.4 0.2 setosa## 2 4.9 3.0 1.4 0.2 setosa## 3 4.7 3.2 1.3 0.2 setosa## 4 5.0 3.6 1.4 0.2 setosa## 5 5.0 3.4 1.5 0.2 setosa## 6 4.4 2.9 1.4 0.2 setosa## 7 4.9 3.1 1.5 0.1 setosa## 8 4.8 3.4 1.6 0.2 setosa## 9 4.8 3.0 1.4 0.1 setosa## 10 4.3 3.0 1.1 0.1 setosa## 11 5.8 4.0 1.2 0.2 setosa## 12 5.7 4.4 1.5 0.4 setosa## 13 5.1 3.5 1.4 0.3 setosa## 14 5.7 3.8 1.7 0.3 setosa## 15 5.1 3.8 1.5 0.3 setosa## 16 4.6 3.6 1.0 0.2 setosa## 17 4.8 3.4 1.9 0.2 setosa## 18 5.0 3.0 1.6 0.2 setosa## 19 5.0 3.4 1.6 0.4 setosa## 20 5.2 3.5 1.5 0.2 setosaCriação de receitas de dados para pré-processamento dos dados
Ambiente de pré-processamento do pacote recipes (Kuhn e Wickham 2021).
Ambiente de pré-processamento do pacote recipes (Kuhn e Wickham 2021).
- Seleção de variáveis
Ambiente de pré-processamento do pacote recipes (Kuhn e Wickham 2021).
Seleção de variáveis
Remoção de valores ausentes
Ambiente de pré-processamento do pacote recipes (Kuhn e Wickham 2021).
Seleção de variáveis
Remoção de valores ausentes
Transformar valores
Ambiente de pré-processamento do pacote recipes (Kuhn e Wickham 2021).
Seleção de variáveis
Remoção de valores ausentes
Transformar valores
etc (ver https://www.tmwr.org/recipes.html#skip-equals-true)
Como funciona?
recipes::recipe(FORMULA, data = NomeDoDataFrame)Ele facilita a criação de "receitas" que permitem a criação e documentação de conjuntos de dados para serem utilizados posteriormente em análises do tidymodels, permitindo ao usuário um fluxo contínuo de análise de dados e a facilidade na reutilização do mesmo conjunto de dados para diversas análises.
Recomendo o aprendizado destas novas (algumas não tão novas, como é o caso do pacote caret ferramentas de pré-processamento e análise de dados em R.
A primeira função desta abordagem é a função recipe(), em que usamos uma fórmula SP1 ~ . para dizer que SP1 é nossa variável resposta e as demais são as variáveis preditoras, representadas pelo . na fórmula.
Porém, sabemos que nosso conjunto de dados dados possui outras variáveis que não são preditoras, como face e coletor.
Uso básico
library("recipes")recipes::recipe(SP1 ~ ., data = dados)#### ── Recipe ─────────────────────────────────────────────────────────────────────────#### ── Inputs## Number of variables by role## outcome: 1## predictor: 1561Aí entra o papel da função update_role() em que informamos o papel de cada variável extra em nosso conjunto de dados.
Por exemplo, as variáveis coletore number são identificadores da coleta.
Então, iniciamos a função com o objeto contendo a receita, dados_receita, e colocamos estas duas variáveis, sem aspas, em seguida, seguido do argumento new_role, em que colocamos um texto informando que papel é esse.
Ao utilizarmos esta receita nos passos de análises, essas variáveis não serão analisadas pois terão a informação desses papéis que elas têm no conjunto de dados.
Atualizando o papel de variáveis extras
Dados do espécime
recipes::recipe(SP1 ~ ., data = dados) %>% update_role(., coletor, number, new_role = "dados do espécime")#### ── Recipe ─────────────────────────────────────────────────────────────────────────#### ── Inputs## Number of variables by role## outcome: 1## predictor: 1559## dados do espécime: 2Face do folíolo
recipes::recipe(SP1 ~ ., data = dados) %>% update_role(., coletor, number, new_role = "dados do espécime") %>% update_role(., face, new_role = "identificador da face do foliolo")#### ── Recipe ─────────────────────────────────────────────────────────────────────────#### ── Inputs## Number of variables by role## outcome: 1## predictor: 1558## dados do espécime: 2## identificador da face do foliolo: 1Identificador do espécime
recipes::recipe(SP1 ~ ., data = treino) %>% update_role(., coletor, number, new_role = "dados do espécime") %>% update_role(., face, new_role = "identificador da face do foliolo") %>% update_role(., especimenid, new_role = "id")#### ── Recipe ─────────────────────────────────────────────────────────────────────────#### ── Inputs## Number of variables by role## outcome: 1## predictor: 1557## dados do espécime: 2## id: 1## identificador da face do foliolo: 1Receita completa
dados_receita <- recipes::recipe(SP1 ~ ., data = treino) %>% update_role(., especimenid, new_role = "id") %>% update_role(., coletor, number, new_role = "dados do espécime") %>% update_role(., face, new_role = "identificador da face do foliolo")dados_receita#### ── Recipe ─────────────────────────────────────────────────────────────────────────#### ── Inputs## Number of variables by role## outcome: 1## predictor: 1557## dados do espécime: 2## id: 1## identificador da face do foliolo: 1Preparo da receita com prep()
dados_receita_prep <- prep(dados_receita)dados_receita_prep#### ── Recipe ─────────────────────────────────────────────────────────────────────────#### ── Inputs## Number of variables by role## outcome: 1## predictor: 1557## dados do espécime: 2## id: 1## identificador da face do foliolo: 1#### ── Training information## Training data contained 36 data points and no incomplete rows.Assando o conjunto teste pós-receita
bake()
teste_processado <- bake(dados_receita_prep, new_data = teste)teste_processado## # A tibble: 12 × 1,562## especimenid face coletor number X10001.03 X3999.64 X4003.497 X4007.354## <int> <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl>## 1 10194 adaxial Perdiz, R.O. 2856 0.258 0.576 0.577 0.577## 2 10196 abaxial Perdiz, R.O. 2858 0.276 0.703 0.703 0.703## 3 10196 adaxial Perdiz, R.O. 2858 0.267 0.712 0.712 0.711## 4 10197 adaxial Perdiz, R.O. 2859 0.274 0.662 0.662 0.661## 5 10197 adaxial Perdiz, R.O. 2859 0.274 0.662 0.662 0.661## 6 10197 adaxial Perdiz, R.O. 2859 0.275 0.710 0.710 0.709## 7 12647 adaxial Perdiz, R.O. 3236 0.320 0.735 0.736 0.736## 8 12648 abaxial Perdiz, R.O. 3237 0.402 0.745 0.746 0.745## 9 12648 abaxial Perdiz, R.O. 3237 0.418 0.748 0.749 0.749## 10 12648 adaxial Perdiz, R.O. 3237 0.389 0.757 0.757 0.757## 11 12648 abaxial Perdiz, R.O. 3237 0.418 0.748 0.749 0.749## 12 12650 abaxial Perdiz, R.O. 3239 0.349 0.733 0.733 0.733## # ℹ 1,554 more variables: X4011.211 <dbl>, X4015.068 <dbl>, X4018.925 <dbl>,## # X4022.781 <dbl>, X4026.638 <dbl>, X4030.495 <dbl>, X4034.352 <dbl>,## # X4038.209 <dbl>, X4042.066 <dbl>, X4045.923 <dbl>, X4049.78 <dbl>,## # X4053.637 <dbl>, X4057.494 <dbl>, X4061.351 <dbl>, X4065.208 <dbl>,## # X4069.065 <dbl>, X4072.922 <dbl>, X4076.779 <dbl>, X4080.635 <dbl>,## # X4084.492 <dbl>, X4088.349 <dbl>, X4092.206 <dbl>, X4096.063 <dbl>,## # X4099.92 <dbl>, X4103.777 <dbl>, X4107.634 <dbl>, X4111.491 <dbl>, …Espremendo os dados treino
juice()
juice(dados_receita_prep)## # A tibble: 36 × 1,562## especimenid face coletor number X10001.03 X3999.64 X4003.497 X4007.354## <int> <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl>## 1 10194 abaxial Perdiz, R.O. 2856 0.278 0.596 0.596 0.596## 2 10194 adaxial Perdiz, R.O. 2856 0.254 0.567 0.567 0.567## 3 10194 abaxial Perdiz, R.O. 2856 0.273 0.581 0.582 0.582## 4 10194 adaxial Perdiz, R.O. 2856 0.258 0.576 0.577 0.577## 5 10194 abaxial Perdiz, R.O. 2856 0.278 0.596 0.596 0.596## 6 10194 adaxial Perdiz, R.O. 2856 0.254 0.567 0.567 0.567## 7 10194 abaxial Perdiz, R.O. 2856 0.273 0.581 0.582 0.582## 8 10196 abaxial Perdiz, R.O. 2858 0.274 0.681 0.681 0.681## 9 10196 adaxial Perdiz, R.O. 2858 0.278 0.673 0.672 0.672## 10 10196 abaxial Perdiz, R.O. 2858 0.274 0.681 0.681 0.681## # ℹ 26 more rows## # ℹ 1,554 more variables: X4011.211 <dbl>, X4015.068 <dbl>, X4018.925 <dbl>,## # X4022.781 <dbl>, X4026.638 <dbl>, X4030.495 <dbl>, X4034.352 <dbl>,## # X4038.209 <dbl>, X4042.066 <dbl>, X4045.923 <dbl>, X4049.78 <dbl>,## # X4053.637 <dbl>, X4057.494 <dbl>, X4061.351 <dbl>, X4065.208 <dbl>,## # X4069.065 <dbl>, X4072.922 <dbl>, X4076.779 <dbl>, X4080.635 <dbl>,## # X4084.492 <dbl>, X4088.349 <dbl>, X4092.206 <dbl>, X4096.063 <dbl>, …Receitas em etapas? Também pode!
# receita para analisedados_receita <- recipes::recipe(SP1 ~ ., data = treino)dados_receita <- update_role(dados_receita, especimenid, new_role = "id")dados_receita <- update_role(dados_receita, coletor, number, new_role = "dados do espécime")dados_receita <- update_role(dados_receita, face, new_role = "identificador da face do foliolo")dados_receita_prep <- prep(dados_receita)dados_receita_prepReceitas em etapas? Também pode!
# receita para analisedados_receita <- recipes::recipe(SP1 ~ ., data = treino)dados_receita <- update_role(dados_receita, especimenid, new_role = "id")dados_receita <- update_role(dados_receita, coletor, number, new_role = "dados do espécime")dados_receita <- update_role(dados_receita, face, new_role = "identificador da face do foliolo")dados_receita_prep <- prep(dados_receita)dados_receita_prepBase R vs. Tidyverse de nosso Curso básico de Introdução ao R para entender o encadeamento (pipeline) do %>%
Vejam que os comandos acima ficaram muito repetitivos, pois a receita dados_receita aparece múltiplas vezes.
Caso utilizássemos o encadeamento de ações proporcionado pelo operador %>% do pacote magrittr (Bache e Wickham 2020), o texto ficaria mais limpo.
Porém, devo admitir que isto é uma questão de preferência.
Normalizar variáveis numéricas
dados_receita_normal <- dados_receita %>% step_normalize(all_predictors())dados_receita_normal#### ── Recipe ─────────────────────────────────────────────────────────────────────────#### ── Inputs## Number of variables by role## outcome: 1## predictor: 1557## dados do espécime: 2## id: 1## identificador da face do foliolo: 1#### ── Operations## • Centering and scaling for: all_predictors()Há várias possibilidades de transformação de dados com as funções step_*()
Há várias possibilidades de transformação de dados com as funções step_*()
https://recipes.tidymodels.org/reference/index.html
Pausa para o café e PERGUNTAS

PCA é um algoritmo matemático que reduz as dimensões dos dados ao mesmo tempo em que mantem a maior parte da variação do conjunto de dados. Ele cumpre essa redução ao identificar direções, chamadas de componentes principais, ao longo das quais a variação dos dados é máxima. Ao utilizar menos componentes, cada amostra pode ser representada por poucas variáveis em oposição à enormidade de variáveis presentes no conjunto de dados brutos. Amostras podem então ser plotadas, permitindo a inferência visual de similaridades e diferenças entre as amostras, e também determinar se as amostras podem ou não ser agrupadas1.
- Ringnér (2008). https://doi.org/10.1038/nbt0308-303.
PCA dentro da receita
dados_receita_pca <- dados_receita %>% step_pca(all_predictors())dados_receita_pca#### ── Recipe ─────────────────────────────────────────────────────────────────────────#### ── Inputs## Number of variables by role## outcome: 1## predictor: 1557## dados do espécime: 2## id: 1## identificador da face do foliolo: 1#### ── Operations## • PCA extraction with: all_predictors()Preparando a PCA
dados_receita_pca_prep <- dados_receita_pca %>% prep()dados_receita_pca_prep#### ── Recipe ─────────────────────────────────────────────────────────────────────────#### ── Inputs## Number of variables by role## outcome: 1## predictor: 1557## dados do espécime: 2## id: 1## identificador da face do foliolo: 1#### ── Training information## Training data contained 36 data points and no incomplete rows.#### ── Operations## • PCA extraction with: X10001.03, X3999.64, X4003.497, X4007.354, ... | TrainedExplorando o resultado com tidy()
pca_tabela <- tidy(dados_receita_pca_prep, 1)pca_tabela## # A tibble: 56,052 × 4## terms value component id ## <chr> <dbl> <chr> <chr> ## 1 X10001.03 -0.0200 PC1 pca_OTwyZ## 2 X3999.64 -0.0444 PC1 pca_OTwyZ## 3 X4003.497 -0.0444 PC1 pca_OTwyZ## 4 X4007.354 -0.0444 PC1 pca_OTwyZ## 5 X4011.211 -0.0444 PC1 pca_OTwyZ## 6 X4015.068 -0.0443 PC1 pca_OTwyZ## 7 X4018.925 -0.0443 PC1 pca_OTwyZ## 8 X4022.781 -0.0442 PC1 pca_OTwyZ## 9 X4026.638 -0.0440 PC1 pca_OTwyZ## 10 X4030.495 -0.0439 PC1 pca_OTwyZ## # ℹ 56,042 more rowsCalculando a variação das PC's
## pcs variacao_porcentagem variacao_cumulativa## 1 PC1 9.989442e+01 99.89442## 2 PC2 1.016427e-01 99.99606## 3 PC3 2.560043e-03 99.99862## 4 PC4 6.865889e-04 99.99931## 5 PC5 3.850244e-04 99.99969## 6 PC6 1.781250e-04 99.99987## 7 PC7 8.591413e-05 99.99996## 8 PC8 1.992217e-05 99.99998## 9 PC9 9.102538e-06 99.99998## 10 PC10 5.233821e-06 99.99999## 11 PC11 3.122542e-06 99.99999## 12 PC12 1.979344e-06 99.99999## 13 PC13 1.600457e-06 100.00000## 14 PC14 8.980482e-07 100.00000## 15 PC15 5.973608e-07 100.00000## 16 PC16 4.585431e-07 100.00000## 17 PC17 4.203126e-07 100.00000## 18 PC18 3.932582e-07 100.00000## 19 PC19 3.433343e-07 100.00000## 20 PC20 2.998497e-07 100.00000## 21 PC21 2.715171e-07 100.00000## 22 PC22 2.340049e-07 100.00000## 23 PC23 1.637715e-30 100.00000## 24 PC24 9.969899e-31 100.00000## 25 PC25 9.969899e-31 100.00000## 26 PC26 9.969899e-31 100.00000## 27 PC27 9.969899e-31 100.00000## 28 PC28 9.969899e-31 100.00000## 29 PC29 9.969899e-31 100.00000## 30 PC30 9.969899e-31 100.00000## 31 PC31 9.969899e-31 100.00000## 32 PC32 9.969899e-31 100.00000## 33 PC33 9.969899e-31 100.00000## 34 PC34 9.969899e-31 100.00000## 35 PC35 9.969899e-31 100.00000## 36 PC36 9.969899e-31 100.00000sdev <- dados_receita_pca_prep$steps[[1]]$res$sdevpercent_variation <- sdev^2 / sum(sdev^2)pca_variacao <- data.frame( pcs = unique(pca_tabela$component), variacao_porcentagem = percent_variation*100, variacao_cumulativa = cumsum(percent_variation*100) )pca_variacaoVisualizando a PCA com ggplot2
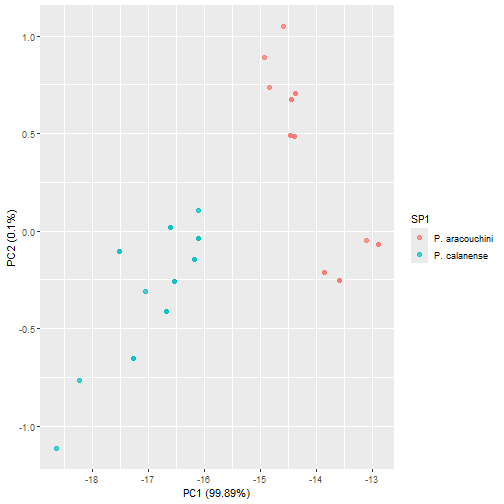
pc1 <- filter(pca_variacao, pcs == "PC1")pc2 <- filter(pca_variacao, pcs == "PC2")juice(dados_receita_pca_prep) %>% ggplot(aes(PC1, PC2)) + geom_point(aes(color = SP1), alpha = 0.7, size = 2) + xlab(paste0("PC1 (", round(pc1$variacao_porcentagem, 2), "%)")) + ylab(paste0("PC2 (", round(pc2$variacao_porcentagem, 2), "%)"))Combinando etapas para a PCA
dados_receita_normal_pca <- dados_receita_normal %>% step_pca(all_predictors()) %>% prep()dados_receita_normal_pca#### ── Recipe ─────────────────────────────────────────────────────────────────────────#### ── Inputs## Number of variables by role## outcome: 1## predictor: 1557## dados do espécime: 2## id: 1## identificador da face do foliolo: 1#### ── Training information## Training data contained 36 data points and no incomplete rows.#### ── Operations## • Centering and scaling for: X10001.03, X3999.64, X4003.497, ... | Trained## • PCA extraction with: X10001.03, X3999.64, X4003.497, X4007.354, ... | TrainedChecando as etapas da PCA com dados normalizados
Dados normalizados
tidy(dados_receita_normal_pca, 1)## # A tibble: 3,114 × 4## terms statistic value id ## <chr> <chr> <dbl> <chr> ## 1 X10001.03 mean 0.309 normalize_0VXeM## 2 X3999.64 mean 0.689 normalize_0VXeM## 3 X4003.497 mean 0.689 normalize_0VXeM## 4 X4007.354 mean 0.689 normalize_0VXeM## 5 X4011.211 mean 0.688 normalize_0VXeM## 6 X4015.068 mean 0.688 normalize_0VXeM## 7 X4018.925 mean 0.687 normalize_0VXeM## 8 X4022.781 mean 0.685 normalize_0VXeM## 9 X4026.638 mean 0.683 normalize_0VXeM## 10 X4030.495 mean 0.681 normalize_0VXeM## # ℹ 3,104 more rowsChecando médias das variáveis
tidy(dados_receita_normal_pca, 1) %>% filter(statistic == "mean")## # A tibble: 1,557 × 4## terms statistic value id ## <chr> <chr> <dbl> <chr> ## 1 X10001.03 mean 0.309 normalize_0VXeM## 2 X3999.64 mean 0.689 normalize_0VXeM## 3 X4003.497 mean 0.689 normalize_0VXeM## 4 X4007.354 mean 0.689 normalize_0VXeM## 5 X4011.211 mean 0.688 normalize_0VXeM## 6 X4015.068 mean 0.688 normalize_0VXeM## 7 X4018.925 mean 0.687 normalize_0VXeM## 8 X4022.781 mean 0.685 normalize_0VXeM## 9 X4026.638 mean 0.683 normalize_0VXeM## 10 X4030.495 mean 0.681 normalize_0VXeM## # ℹ 1,547 more rowsChecando desvio padrão das variáveis
tidy(dados_receita_normal_pca, 1) %>% filter(statistic == "sd")## # A tibble: 1,557 × 4## terms statistic value id ## <chr> <chr> <dbl> <chr> ## 1 X10001.03 sd 0.0417 normalize_0VXeM## 2 X3999.64 sd 0.0601 normalize_0VXeM## 3 X4003.497 sd 0.0601 normalize_0VXeM## 4 X4007.354 sd 0.0602 normalize_0VXeM## 5 X4011.211 sd 0.0602 normalize_0VXeM## 6 X4015.068 sd 0.0601 normalize_0VXeM## 7 X4018.925 sd 0.0601 normalize_0VXeM## 8 X4022.781 sd 0.0600 normalize_0VXeM## 9 X4026.638 sd 0.0599 normalize_0VXeM## 10 X4030.495 sd 0.0597 normalize_0VXeM## # ℹ 1,547 more rowsChecando a PCA normalizada
pca_tabela_normalizada <- tidy(dados_receita_normal_pca, 2)pca_tabela_normalizada## # A tibble: 56,052 × 4## terms value component id ## <chr> <dbl> <chr> <chr> ## 1 X10001.03 0.0258 PC1 pca_lZPx9## 2 X3999.64 0.0222 PC1 pca_lZPx9## 3 X4003.497 0.0222 PC1 pca_lZPx9## 4 X4007.354 0.0223 PC1 pca_lZPx9## 5 X4011.211 0.0223 PC1 pca_lZPx9## 6 X4015.068 0.0223 PC1 pca_lZPx9## 7 X4018.925 0.0224 PC1 pca_lZPx9## 8 X4022.781 0.0224 PC1 pca_lZPx9## 9 X4026.638 0.0224 PC1 pca_lZPx9## 10 X4030.495 0.0224 PC1 pca_lZPx9## # ℹ 56,042 more rowsCalculando a variação das PC's
## pcs variacao_porcentagem variacao_cumulativa## 1 PC1 9.443263e+01 94.43263## 2 PC2 5.335957e+00 99.76858## 3 PC3 1.317963e-01 99.90038## 4 PC4 5.227559e-02 99.95265## 5 PC5 2.774673e-02 99.98040## 6 PC6 1.328499e-02 99.99369## 7 PC7 3.438648e-03 99.99712## 8 PC8 9.002714e-04 99.99802## 9 PC9 7.762045e-04 99.99880## 10 PC10 4.287108e-04 99.99923## 11 PC11 2.452045e-04 99.99947## 12 PC12 1.455485e-04 99.99962## 13 PC13 9.697651e-05 99.99972## 14 PC14 5.609298e-05 99.99977## 15 PC15 4.162806e-05 99.99982## 16 PC16 3.964418e-05 99.99985## 17 PC17 3.855582e-05 99.99989## 18 PC18 3.353950e-05 99.99993## 19 PC19 2.809163e-05 99.99995## 20 PC20 2.407041e-05 99.99998## 21 PC21 2.097662e-05 100.00000## 22 PC22 3.603135e-29 100.00000## 23 PC23 2.349727e-30 100.00000## 24 PC24 8.521586e-31 100.00000## 25 PC25 8.521586e-31 100.00000## 26 PC26 8.521586e-31 100.00000## 27 PC27 8.521586e-31 100.00000## 28 PC28 8.521586e-31 100.00000## 29 PC29 8.521586e-31 100.00000## 30 PC30 8.521586e-31 100.00000## 31 PC31 8.521586e-31 100.00000## 32 PC32 8.521586e-31 100.00000## 33 PC33 8.521586e-31 100.00000## 34 PC34 8.521586e-31 100.00000## 35 PC35 8.521586e-31 100.00000## 36 PC36 8.521586e-31 100.00000sdev_pca_normalizada <- dados_receita_normal_pca$steps[[2]]$res$sdevporcent_variacao_normalizada <- sdev_pca_normalizada^2 / sum(sdev_pca_normalizada^2)pca_normalizada_variacao <- data.frame( pcs = unique(pca_tabela_normalizada$component), variacao_porcentagem = porcent_variacao_normalizada*100, variacao_cumulativa = cumsum(porcent_variacao_normalizada*100) )pca_normalizada_variacaoVisualizando a PCA com dados normalizados
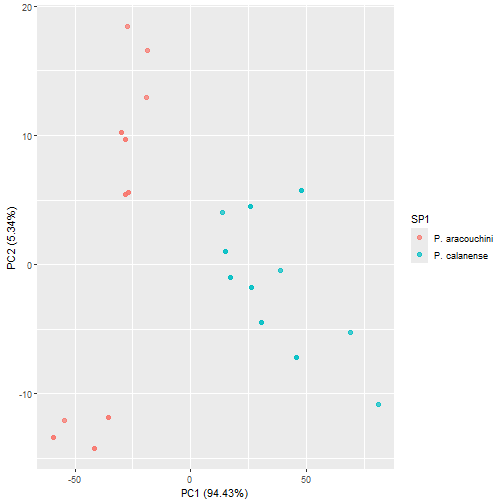
pc1_normal <- filter(pca_normalizada_variacao, pcs == "PC1")pc2_normal <- filter(pca_normalizada_variacao, pcs == "PC2")juice(dados_receita_normal_pca) %>% ggplot(aes(PC1, PC2)) + geom_point(aes(color = SP1), alpha = 0.7, size = 2) + xlab(paste0("PC1 (", round(pc1_normal$variacao_porcentagem, 2), "%)")) + ylab(paste0("PC2 (", round(pc2_normal$variacao_porcentagem, 2), "%)"))PERGUNTAS?

Referências importantes
Kuhn, M. e Johnson, K. 2019. Feature Engineering and Selection: A Practical Approach for Predictive Models. CRC Press.
Kuhn, M. 2019. The caret package. Disponível em https://topepo.github.io/caret/index.html.
Kuhn, M. 2019. A Short Introduction to the caret Package. Disponível em https://cran.r-project.org/web/packages/caret/vignettes/caret.html.
Kuhn, Max e Silge, J. 2021. Tidy Modeling with R. Disponível em https://www.tmwr.org/base-r.html.
Perdiz, R. O que é o tidyverse? Disponível em https://www.ricardoperdiz.com/blog/2020-04-tidyverse/
Silge, Julia. Todas suas postagens são úteis. #tidytuesday #tidymodels.
Wickham, H. e Grolemund, J. 2017. R for Data Science. Disponível em https://r4ds.had.co.nz.